Espaço para compartilhar e colaborar com scripts no Google Earth Engine
Olá pessoal!
Fiz um script no GEE para baixar o mapa de uso e cobertura do solo de 2017, derivada da coleção 3, para o bioma da Caatinga. Venho aqui compartilhar.
///// importar mapas de uso da coleção 3 - 2017
var image = ee.Image(‘projects/mapbiomas-workspace/public/collection3/mapbiomas_collection3_integration_v1’);
Map.addLayer(image,{min:0,max:100,bands:[‘classification_2017’]},‘Uso_2017’);
/// importar tabela com shapefile da Caatinga
Map.addLayer (table,{},‘Caatinga’,true);
///// exportar recorte do uso 2017 para a Caatinga
Export.image.toDrive({
image:image.select([‘classification_2017’]),
fileNamePrefix: ‘Caat_uso_2017’,
description:‘raster’,
folder:‘MapBiomas2017’,
region: table,
scale:30,
maxPixels:10000000000});
Olá Adriana. Troque a parte da simbologia pelo código abaixo:
// Require the Palettes module
var palettes = require(‘users/mapbiomas/modules:Palettes.js’);
var visclass3 = {
_ ‘bands’: ‘classification_2017’,_
_ ‘min’: 0,_
_ ‘max’: 33,_
_ ‘palette’: palettes.get(‘classification2’),_
_ ‘format’: ‘png’_
_ }_
Deve melhorar a visualização das cores do mapa.
Olá,
tenho pouca experiência com Engine e tentei fazer um scrip para calcular a proporção de cada classe em um shapefile com diferetens feições (ex municipios ou bacias). Porém tanto exportando em CSV e GSON as proporções (entre 0 e 1) de cada classe não aparecem em colunas, mas sim todas as classes como texto em uma mesma coluna. Por exemplo, a coluna proportion para uma geometria aleatória com 3 feições retorna assim:
system:index; proportion
0 {33=0.007666061035882462, 0=6.39561097250995E-6, 12=0.03430513100623398, 24=2.171375186503582E-4, 13=0.006537258681461959, 3=0.7613429647558654, 25=5.921474822756637E-4, 4=0.03999446690446732, 15=0.10365565783493545, 27=1.2520963234056694E-6, 10=5.742997607968119E-7, 21=0.045680938674588806};
1 {33=0.001046649654060303, 0=2.8282300647823665E-5, 12=0.21683808907995594, 24=3.857948838235778E-4, 3=0.08825521543938591, 4=0.16660015993576538, 15=4.539455132055185E-4, 19=0.08386999103479202, 21=0.44252190005811504};
2 {33=0.02794267706211883, 12=0.013064444257980666, 24=0.005448399300853789, 25=9.57615225618418E-4, 15=0.45412612143320064, 19=0.02456674341005589, 0=1.3492630166997554E-5, 3=0.06500602711685988, 4=0.011920404813896284, 9=4.207270113760412E-5, 30=2.5347426614301763E-5, 20=0.11033325953380718, 21=0.28655340415185726}
Creio que não seja algo dificil, mas até aqui não deu certo.
Segue o script
var mb = ee.Image(“projects/mapbiomas-workspace/public/collection3/mapbiomas_collection3_integration_v1”),
var munic = ee.FeatureCollection(“projects/mapbiomas-workspace/AUXILIAR/municipios-2016”);
// Select one band as example
var yr = mb.select(‘classification_1985’);
// Extract the values with histogram
var frequency = yr.reduceRegions({
collection: munic,
reducer: ee.Reducer.frequencyHistogram(),
scale: 30
});
// transform histogram to proportion
var propLUC = function(feature) {
var dict = ee.Dictionary(feature.get(‘histogram’));
var sum = ee.Array(dict.values()).reduce(ee.Reducer.sum(),[0]).get([0]);
var proportion = dict.map(function(k,v) {
return ee.Number(v).divide(sum);
});
return feature.set(‘proportion’, proportion);
};
//Map over features
var LUCprop = frequency.map(propLUC);
// Export results
Export.table.toDrive({
collection: ee.FeatureCollection(LUCprop),
description:‘teste’,
fileFormat: ‘CSV’
});
Olá João, boa tarde
Primeiro obrigado pela utilização dos dado MapBiomas e do Fórum para sanar suas dúvidas.
Antes de lhe passar e explicar o código corrigido, sugiro que compartilhemos nossos scripts usando o botão “Get Link” e colando aqui o endereço que aparecerá no seu browser. Isso deixará as coisas mais enxutas e fáceis de corrigir.
Vamos ao seu código…O código em si faz sentido, o que temos aqui é uma mensagem de erro pouco precisa do GEE. O que dificulta a bastante a solução do problema. Na sua tentativa de encontrar a proporção das classes, dentro de geometrias previamente definidas, há uma geometria que está sob área sem imagem. Trata-se de parte do arquipélago de Fernando de Noronha.
A correção está disponível aqui:
https://code.earthengine.google.com/1c3ae3ce7d8322ce5bc3b008230c346f
A solução passa por filtrar essa região, excluindo-a da análise (ver linha 23)
Outra coisa é que todos os municípios postos juntos, podem lhe gerar uma mensagem de “timeout” durante a exportação do dado. Assim, tomei a liberdade de quebra-lo em pedaços, usando ID de das UF’s Brasileiras, veja as linhas 7 até 17 e a linha 58.
Espero ter ajudado,
Att
Equipe Solved
Olá Cesar,
Obrigado pela resposta e desculpe pela desatenção no modo de compartilhamento.
De fato esse código era o que eu estava buscando.
Com relação à exportação das proporções em string e não em colunas, consigo solucionar no proprio CSV. Creio que para fazer isso direto do GEE precisaria criar cada feição com os valores de raster (0-33) e ir preenchendo cada coluna. Mas esse script já atende à minha demanda.
Muito obrigado!
Abs
Boa tarde, Cesar!
Por favor, como faço para unir o raster do Mapbiomas (download pelo GEE) com a planilha do excel? Obrigada!
Olá pessoal.
Sou bem iniciante no GEE, então ainda não tive sucesso em resolver uma questão que pensei ser útil: como habilitar uma opção em que possamos escolher visualizar a mudança da cobertura do solo entre um par de anos a nossa escolha e não apenas as datas pré-definidas?
Obrigado
Olá Davi,
O cálculo de transições e geração de estatísticas é demorado e tem que ser executado em BATCH. Nos mapas de transições já deixamos pré-calculado os períodos do inventário florestal, código florestal, primeiro e último ano, 5 em 5 anos e anos subsequentes (http://mapbiomas.org/map#transitions). Para outros períodos, as transições devem ser geradas pelos mapas de cobertura, que estão disponíveis no GEE:
id: projects / mapbiomas-workspace / public / collection3_1 / mapbiomas_collection31_integration_v1
Bom dia, pessoal.
Pelo que entendi aqui no fórum, o que for relativo a script deve vir para cá. Não sei se minha pergunta se enquadra aqui exatamente, mas aí vai. São algumas, na verdade.
Estou baixando os rasters que vocês usaram e estão disponíveis, usando essa postagem aqui, do Tasso: https://l.facebook.com/l.php?u=http%3A%2F%2Fforum.mapbiomas.discourse.com.br%2Ft%2Fcomo-baixar-mosaicos-landsat-do-mapbiomas%2F53%3Ffbclid%3DIwAR0gX1Vdkw0fsJwAvUTSD1kI9178bF5UyPx3BI2V1lVcgy8uYtpsIWyNYcQ&h=AT3oztoGrhKUI6f1ke6cyXHOjm4f_eRcf5IbnYuq-uUiqeBLNaMN0-k-HQG43H7BW8gZlLXMLoLEWOmr4XP0EdT26713tv86SHSQChPX11ygr16hbRtU_fccfnAaHEGfzq_7MQ
Bem, adaptei o código para o que eu queria e ele está aqui: https://l.facebook.com/l.php?u=http%3A%2F%2Fforum.mapbiomas.discourse.com.br%2Ft%2Fcomo-baixar-mosaicos-landsat-do-mapbiomas%2F53%3Ffbclid%3DIwAR0gX1Vdkw0fsJwAvUTSD1kI9178bF5UyPx3BI2V1lVcgy8uYtpsIWyNYcQ&h=AT3oztoGrhKUI6f1ke6cyXHOjm4f_eRcf5IbnYuq-uUiqeBLNaMN0-k-HQG43H7BW8gZlLXMLoLEWOmr4XP0EdT26713tv86SHSQChPX11ygr16hbRtU_fccfnAaHEGfzq_7MQ
Minhas perguntas são:
- Tem como, na parte onde especifico as coordenadas, eu importar um polígono com diversos pontos, para eu não ter que ficar digitando um por um?
- Essas imagens que baixei já são NDVI e EVI (medianas) calculados, pelo que entendi, certo? Como foram calculados o NDVI e EVI? Ainda sobre NDVI e EVI: são a mediana por pixel das várias imagens que vocês tem ao longo do ano (por exemplo, se eu pedir lá o ano de 2015, o raster resultante será em que cada pixel será o valor mediano do NDVI e EVI ao longo do ano)?
- Como faço para var quais bandas estão disponíveis para serem baixadas?
Tenho outras perguntas, mas talvez eu consiga resolver com base nas que estão acima.
Obrigado!
Thiago
Oi Thiago,
Seguem as respostas.
1. Tem como, na parte onde especifico as coordenadas, eu importar um polígono com diversos pontos, para eu não ter que ficar digitando um por um?
Esse script exporta a imagem dentro de uma “geometry”. Você pode importar um shapefile como “Asset” e depois importá-lo como “FeatureCollection” e utilizá-lo no lugar da geometry.
2. Essas imagens que baixei já são NDVI e EVI (medianas) calculados, pelo que entendi, certo? Como foram calculados o NDVI e EVI?
Os códigos abaixo utiliza a função “.expression”. É importante lembrar que também existe uma função “.normalizedDifference” que também pode ser utilizada.
var ndvi = image.expression(‘float(nir - red)/(nir + red)’, {
‘nir’: image.select(‘nir’),
‘red’: image.select(‘red’)
}).multiply(100).byte();
image = image.addBands(ndvi.select([0],[‘NDVI’]));
var evi2 = image.expression(‘float(2.5 * (nir - red)/(nir + 2.4 * red + 1))’, {
‘nir’: image.select(‘nir’),
‘red’: image.select(‘red’),
}).multiply(100).byte();
image = image.addBands(evi2.select([0],[‘EVI2’]));
Ainda sobre NDVI e EVI: são a mediana por pixel das várias imagens que vocês tem ao longo do ano (por exemplo, se eu pedir lá o ano de 2015, o raster resultante será em que cada pixel será o valor mediano do NDVI e EVI ao longo do ano)?
Essas bandas são a mediana do NDVI e EVI2 de todos os pixels válidos (removidos ruídos, nuvem e sombra de nuvem) do melhor período do ano escolhido para cada Bioma. O ATBD de cada bioma indica qual o período que foi utilizado por padrão, que pode variar entre 3 a 6 meses na média, normalmente no final da época chuvosa e durante época seca. (https://mapbiomas.org/atbd-3)
3. Como faço para var quais bandas estão disponíveis para serem baixadas?
Você pode adicionar a função “print(image)” na linha 9 (logo após a linha “image = image.mosaic();”).
Esse código vai mostrar a imagem com as 102 bandas disponíveis no console. Qualquer uma dessas bandas pode ser exportada.
Veja que na linha 11 estão selecionadas quais bandas vão ser exportadas (“image:image.select([‘median_ndvi’,‘median_evi2’]),”. Você pode alterar aí para qualquer uma das 102 bandas disponíveis.
Um abraço,
Marcos
Muito obrigado pela resposta, Marcos! E desculpe a demora para responder. Eu acompanhei diariamente por uns dias, mas estava com muitas abas abertas na internet e fechei a de vocês esperando que o google me alertasse quando houvesse resposta, mas ele não o fez hehehe…
Vou dar uma fuçada por aqui e qualquer coisa, entro em contato de novo.
Abraços!
Olá Marcos, eu de novo! hehehe…
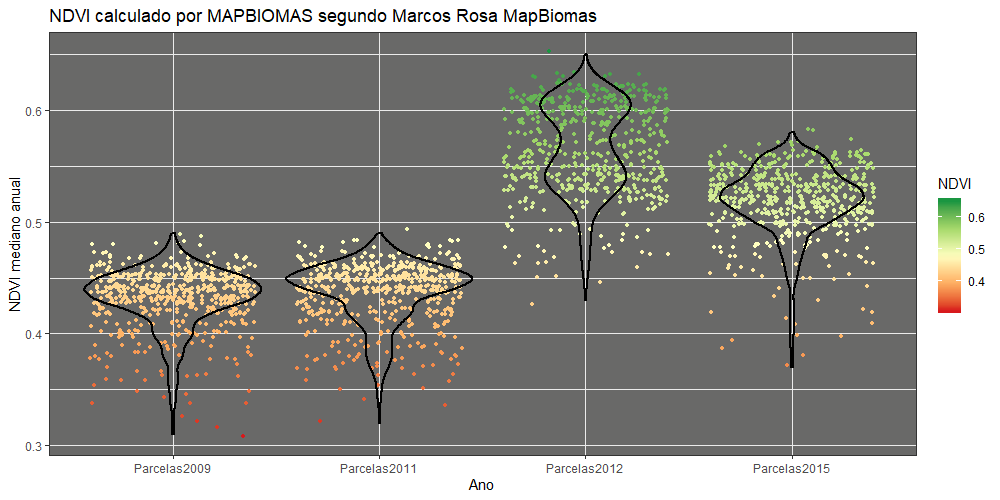
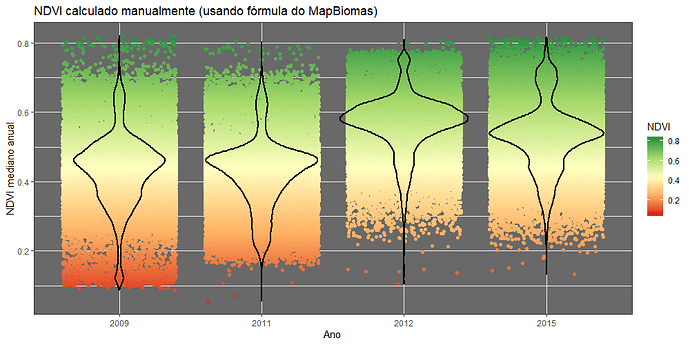
Eu baixei as bandas separadamente (mediana anual) da área que me interessa e fiz o cálculo do NDVI com a fórmula que você informou acima com o R ((nir - red)/(nir + red)). Depois disso eu peguei o que baixei do banco de dados de vocês mesmo, já pronto (banda median_ndvi). Os resultados seguem mais ou menos o mesmo padrão de mabas as formas, mas as diferenças ficam muito menos evidentes usando o que vocês disponibilizam já calculado. O número de ponto é bem menor usando o que vocês disponibilizaram e eu imagino que por isso haja essa diferença. Escrevo para perguntar o motivo de o número de pontos diminuir, é por causa das correções? Retirada de nuvens? Essas perguntas não são de maneira nenhuma querendo contestar nada, eu só quero entender mesmo! Aviso porque nesses tempos de contestação de tudo (principalmente de dados científicos) pode ser importante deixar claro hehe.
Coloquei as imagens abaixo, para você ver o que estou falando.
Abraços,
Thiago
Oi Thiago. O nosso script calcula o NDVI de cada imagem Landsat e depois gera a mediana do NDVI. Pelo que entendi, no seu caso, você calculou o NDVI das bandas de Mediana do NIR e RED.
Pelo que entendi, você está comparando a banda median_NDVI do mosaico do nosso projeto e uma banda de mediana do NDVI que você calculou em R usando a median_NIR e median_RED do mosaico que disponibilizados. A diferença na quantidade de pontos nos gráficos não pode ser justificada por essa descrição. Tem alguma diferença aí que não consegui entender. Essa diferença tão grande de pontos só seria justificada se você estivesse comparando com o NDVI de cada imagem e não com a mediana do ano.
Outra sugestão para você testar, que pode ser interessante são as bandas “median_ndfi_dry” e “median_ndfi_wet”
Bom dia
Estou tentando baixar imagens landast pra acompanhamento de áreas embargadas. Mas o script que uso tá apresentado dois erros. Alguém poderia me auxiliar na correção?